本期分享一篇关于宿主微生物与结直肠癌(CRC)相关性的文章,主要描述了CRC 中每个共识分子分型(CMS)的肿瘤微环境图谱,包括宿主基因和瘤内微生物组。文章于2023年12月发表于《Physiological Genomics》,通讯作者为浙江大学的焦娜教授,标题“Host-microbiota interactions contributing to the heterogeneous tumor microenvironment in colorectal cancer”。
文章亮点
1、研究系统地描绘了结直肠癌(CRC)中不同共识分子分型(CMS)特异性免疫图谱。
2、研究首次进行了CMS特异性基因-微生物关联性分析,揭示了每个CMS中不同的相互作用模式。
1 研究背景
结直肠癌(CRC)是全球最常见的癌症之一,由于CRC患者遗传、表观遗传和TME的变化,其治疗反应也不尽相同。
为了更好地对CRC患者进行分层,国际结直肠癌亚型联盟制定了共识分子分型(CMS),描述了四种具有分子和临床差异的共识分子亚型:CMS1(微卫星不稳定型,MSI)、CMS2(经典型)、CMS3(代谢型)和CMS4(间质型)。
这种分类有助于更精准地了解不同亚型的肿瘤生物学特征,为个体化治疗和临床决策提供了更多信息。
2 研究内容
1. 从TCGA数据库过滤CRC样本,分析宿主基因表达谱提示不同CMS间差异较大。
2. 免疫浸润分析提示,CMS1种CD8+T细胞高浸润,CMS4中以M0和M2巨噬细胞为主。
3. 肠道微生物共丰度网络提示CMS1与微生物关联复杂,有更强的免疫激活状态。
4. 相关性分析发现,不同CMS中的关联微生物有各自对应的铁死亡相关基因。
3 研究结果
01基于宿主基因表达的CMS特征
为了全面探讨CRC中不同CMS的TME特征,作者对宿主基因表达模式、瘤内微生物特征进行了关联性分析。首先,从TCGA数据库中提取了总共594个具有匹配的宿主基因表达谱和瘤内微生物丰度谱的CRC样本。
作者通过分析宿主基因表达谱,发现了大量差异基因,提示了CMS之间的巨大异质性。作者进一步发现铁死亡相关基因的表达水平在不同CMS之间差异很大,没有发现共同的驱动基因和抑制基因。其中,铁死亡在CMS3中受到抑制,但在CMS1和CMS4中相对活跃(图1)。
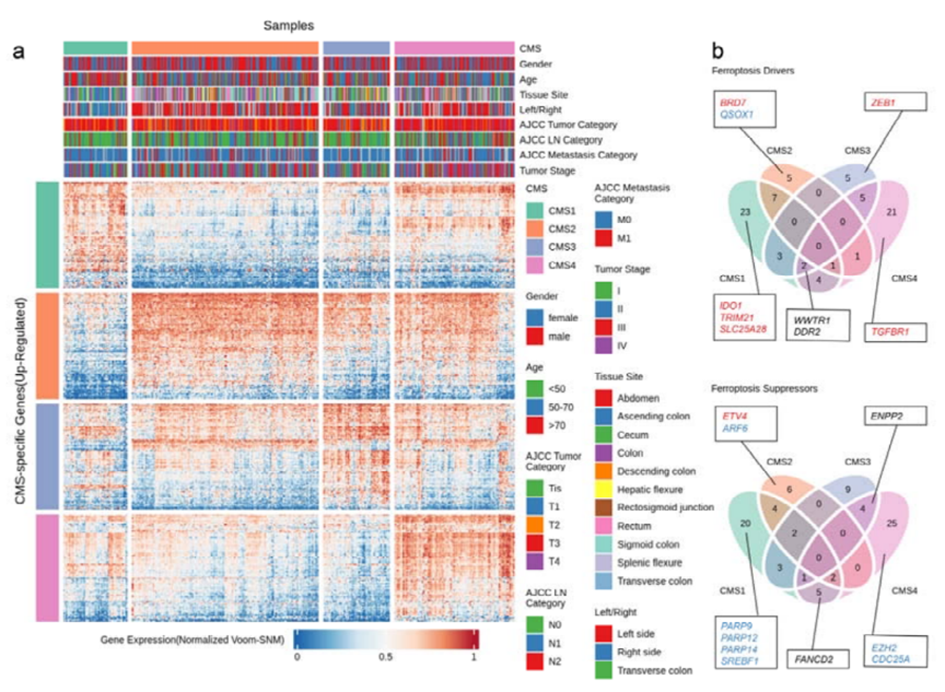
图1:CMS特异性的宿主基因表达
02 CRC中不同CMS的免疫浸润分析
随后,作者对每类CMS的差异基因进行通路富集分析,结果提示CMS1和CMS4显示出比CMS2和CMS3更高的免疫评分。CMS1表现出更高水平的CD8+T细胞、滤泡辅助性T细胞和M1巨噬细胞,同时Treg细胞和单核细胞的浸润水平下降。
相比之下,CMS4以M0和M2巨噬细胞为主,M1巨噬细胞和CD8+T细胞较少,这也提示CMS4中炎性成分的浸润较低(图2)。
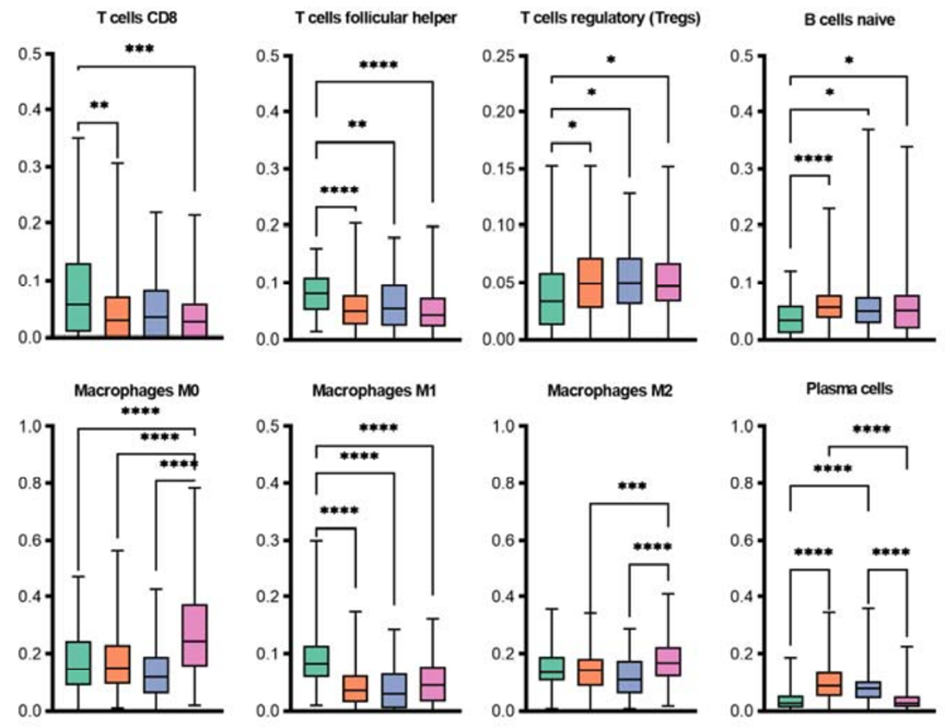
图2:不同CMS的免疫浸润分析
03 不同CMS的肠道微生物群特征
尽管与邻近正常组织相比,CRC肿瘤组织中的微生物α多样性有所降低。作者进一步分析了不同CMS中的肠道菌群特征,其中变形菌门是占CRC组织中的优势门,而粪便中的优势门,如厚壁菌门和拟杆菌门却占比不大。
其中,CMS1与60个属的表现出更复杂的关联,这与其肠道微生物群参与的强免疫激活状态一致(图3)。这也证实了CMS中瘤内微生物环境的不同结构和关键属,表明瘤内微生物在TME异质性中的潜在贡献。
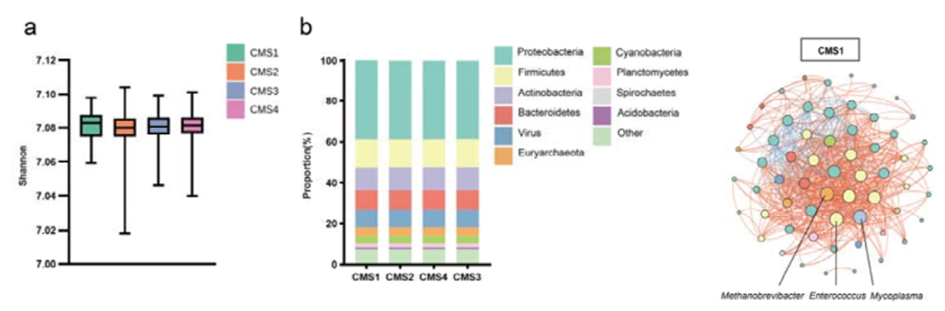
图3:CMS微生物群的特征
04 CMS中宿主-微生物群的相互作用
受CMS中铁死亡的独特状态的启发,作者关注了铁死亡相关的基因与微生物之间的联系。
· 在CMS1中,火色杆菌属与铁死亡驱动基因DDR2呈正相关。
· 在CMS2中,Apibacter(一类拟杆菌)与驱动基因PGRMC1呈正相关,与抑制基因SRC呈负相关。
· 在CMS3中,瘤胃球菌属与抑制基因RARRES2之间呈正相关,管胞藻属与驱动基因ACSL4以及IDH1之间呈负相关。
· 在CMS4中,大多数关联发生在萨特氏菌属之间,包括驱动基因DDR2、WWTR1和TIMP1,表明萨特氏菌在CMS4中铁死亡失调中发挥重要作用。
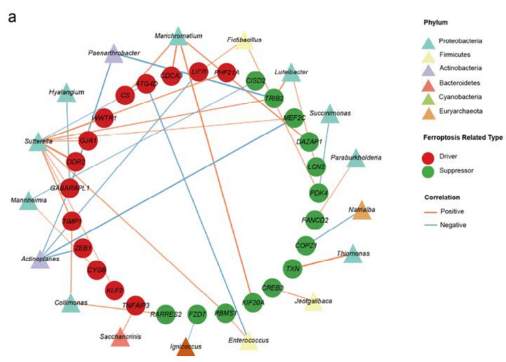
图4:CMS中与铁死亡相关的基因微生物关联
4 文章总结
该研究通过TCGA过滤的594个CRC样本的转录组和微生物群落,鉴定出不同CMS之间的基因和微生物差异,并评估宿主-微生物群相互作用对TME的影响,为阐明CRC异质性机制和潜在治疗靶点提供了新思路。
不过本文也有以下不足:
1、样本数量有限:该研究只使用了594个CRC样本进行分析,这在某种程度上可能限制了结果的可靠性和泛化能力。
2、数据来源局限性:该研究的宿主转录组和瘤内微生物组数据源自TCGA的RNA-seq数据,缺乏临床的自测数据是本文的一大弱点。
3、结果的功能解释不足:尽管该研究提供了基因和微生物群的差异,以及它们与TME的相互作用,但在功能解释方面缺乏详细的说明。
5 拓展研究
本文对每个CMS中的基因-微生物关联进行了分析,并揭示了不同CMS中的相互作用模式。但是这个结论放在基金的申请上,论证强度还是不够的,我们可以从以下几个方面着手加强:
1、进一步探索这些关联的具体机制,例如通过分析代谢途径和免疫调节等方面的功能解释,以深入了解不同CMS之间的差异。
2、通过针对特定CMS类型中的微生物或宿主基因进行干预,可能有助于提高肿瘤治疗的效果,为肠癌的治疗和管理提供更准确的指导。
3、通过采用组学和系统生物学方法,深入研究基因-微生物关联的机制,例如通过建立基于微生物代谢物和宿主代谢物的互作网络来阐明其功能。













